在全球范围内,肝细胞癌(hepatocellular carcinoma,HCC)是常见的癌症之一,在因癌症死亡原因中占第4位,并且其发病率预计将来还会增加[1]。HCC的主要治疗手段为肝癌切除术[2],而手术后较高的转移率和复发率严重影响着HCC患者的治疗效果和生存期[3-4]。且由于HCC早期无明显症状难以发现、较早出现肝内转移等原因导致许多HCC患者在诊断时已经失去了手术的机会[5]。因此有效的预后标志模型可能有助于指导个体治疗策略的制定从而改善患者生存期。
自噬是溶酶体依赖细胞内降解途径,通过降解不需要的细胞成分在生理、病理生理环境中对细胞生存、分化、发育和调节细胞稳态至关重要的生理过程[6]。在生理情况下,自噬通过细胞内降解机制分解受损的细胞器从而维持细胞内环境稳定[7]。自噬过程同样参与许多病理过程,如神经退行性疾病,衰老和癌症[8-9]。越来越多的证据表明自噬失调与肝脏疾病如脂肪肝、肝硬化和HCC的发病机理有关[10]。近些年,许多研究小组试图将自噬基因确立为癌症的潜在治疗靶标[11-12]。有研究[13-14]表明自噬药物可以用作药理剂,以克服HCC治疗中化疗的局限性,例如药物毒性和耐药性。但这是否是一个替代性的和正在出现的概念仍然是一个巨大的挑战[15]。以自噬调节为视角和潜在治疗目标的研究数目越来越多,自噬可能将成为一种潜在的方法来治疗HCC[16]。有研究[17]表明IFN-γ通过诱导自噬体的形成和微管相关蛋白1轻链3蛋白的转化抑制人HCC细胞生长和非凋亡性细胞死亡。越来越多的研究突显了自噬在HCC中的重要性,其中确定主要的自噬靶标研究尤为重要。
长链非编码RNA(long non-coding RNA,lncRNA)是一类无蛋白编码功能的RNA序列,lncRNA涉及RNA衰变,基因表达的遗传调控,RNA剪接,microRNA调控和蛋白质折叠等多种生理过程[18]。其异常表达与多种癌症的发生、发展密切相关,并具有诊断及预后价值[19]。通过lncRNA-mRNA共表达分析可以较好评估lncRNA的功能[20-21]。在HCC发生、发展及预后中,发现许多lncRNA在扮演着重要作用。有研究[22-23]表明,在HCC中存在lncRNA介导转录和自噬相关基因转录后水平调控自噬调控网络。因此,自噬相关的lncRNA可能在HCC患者的预后中具有潜在价值,并且可以作为潜在的治疗靶标。本研究旨在建立HCC中自噬相关的lncRNA预后模型。
1 材料与方法
1.1 材料
HCC患者转录组测序及临床数据从TCGA数据率获取(https://cancergenome.nih.gov)。下载的转录组测序数据包含HCC患者374例癌组织以及50例癌旁组织的mRNA和lncRNA表达数据。HCC患者的临床数据包括374例HCC的临床相关资料,如生存时间、生存状态、年龄、性别、肿瘤分级和病理分期等。
1.2 方法
1.2.1 自噬相关lncRNA 的筛选 从HADb 网站(http://autophagy.lu/clustering/index.html)获 取自噬基因列表。从374 例HCC TCGA RNA 数据集中获得lncRNA 和自噬相关基因的表达数据。利用Pearson 相关性分析计算lncRNA 与自噬相关基因的相关性。满足|R2|>0.3 且P<0.001 的lncRNA 被认为是自噬相关的lncRNA。
1.2.2 数据的预处理 删除在HCC 患者中生存时间少于30 d 的患者,用于生存分析的数据集包括342 例HCC 患者的自噬相关lncRNA 表达数据和临床资料。利用R 软件中caret 程序包将用于预后分析的342 例TCGA 的HCC 数据集样本按70% 与30% 的比例分为训练集(n=242)和内部验证集(n=100)。
1.2.3 风险预后模型的构建及分析 首先,进行单因素Cox 回归分析及KM 法以评估自噬相关lncRNA 的预后价值。筛选出满足上述2 种方法P 值均<0.05 与HCC 患者预后相关的lncRNA。使用多因素Cox 回归分析对具有预后意义的自噬相关lncRNA 进行筛选并建立风险评分模型。使用风险评分计算公式为每个患者计算风险评分:风险评分=coef基因1×expr基因1+coef 基因2×expr 基因2+ ...+coef基因Ñ×expr基因Ñ。通过加权lncRNA 的表达水平与回归系数(coef)得到风险评分。coef值是通过多因素Cox 回归分析得到的回归系数;lncRNA 表达定义为expr 基因n。根据中位风险评分划分高风险组和低风险组。利用R 3.6.3 软件中survival 程序包绘制模型预后的生存曲线,并比较两组患者之间生存的差异。绘制预后模型的ROC曲线以评价模型的灵敏度和特异性。使用R 软件中survivalROC 程序包绘制ROC 曲线并计算其AUC 数值。
1.3 统计学处理
使用Kaplan-Meier法比较高风险组和低风险组的总生存期(OS),采用对数秩检验计算P值。使用单变量和多变量Cox分析来评估风险评分与临床病理特征或OS之间的关系。P<0.05为差异有统计学意义。自噬相关lncRNA共表达网络的构建使用Cytoscape软件(版本3.6.0)完成。
2 结 果
2.1 自噬基因-lncRNA 共表达网络的构建
从TCGA数据库中提取了TCGA数据集中总共 3888个lncRNA。从HADb中总共提取了256个自噬相关基因。通过构建自噬相关基因-lncRNA共表达网络,鉴定与自噬相关的lncRNA。最后,347个lncRNA鉴定为自噬相关lncRNA。
2.2 鉴定HCC 患者的12 种自噬相关lncRNA 的特征
在242例TCGA训练集中基于347种自噬相关的lncRNA使用单因素Cox回归分析和KM法来筛选具有预后意义的自噬相关lncRNA。同时满足Kaplan-Meier法及单因素Cox方法P值均<0.05的lncRNA被考虑为具有预后意义的自噬相关lncRNA。共有26个lncRNA对HCC患者具有预后价值(P<0.05)。随后,进一步使用多因素Cox回归分析对这些lncRNA进行筛选后构建预后模型,同时依据赤池信息量准则(Akaike information criterion,AIC)对模型进行优化,最优AIC=797.82。最后12个lncRNA被用于构建预后模型(表1)(图1)。随后构建预后相关自噬lncRNA共表达网络(图2)。同时绘制12个自噬相关lncRNA和自噬相关基因及对患者总生存期的关系的组合桑基图(图3)。
2.3 自噬相关的lncRNA 模型对HCC 的预后影响
使用风险评分公式计算自噬相关lncRNA模型的风险评分(表2),风险评分公式:风险分数= coef基因1×expr基因1+coef基因2×expr基因2+...+coef基因Ñ×expr基因Ñ。按中位风险评分值将训练集HCC患者分为两组(低风险组和高风险组)。利用R 3.6.3软件中的survival程序包根据患者风险评分分析生存差异,绘制风险评分曲线、生存状态图及12个lncRNA表达热图(图4)。随着风险评分的升高,高风险评分组患者的死亡人数显著多于低风险评分组,说明高风险评分组的患者整体生存率较差。风险模型还可以预测HCC患者的生存时间,其中低风险组的总生存时间比高风险组的总生存时间更长(图5)。此外,还通过多元Cox回归分析探讨了风险评分签名是否是HCC患者预后的独立预测因子。在消除了其他因素(如性别,年龄,肿瘤分级和分期)的影响后,该风险评分模型仍可显著预测HCC患者的生存(HR=1.201,95% CI=1.141~1.264,P<0.001)(表3)。随后,利用survivalROC程序包绘制模型的ROC曲线以评价模型的灵敏度和特异性(1、3和5年AUC分别为0.801,0.819和0.787)(图6)。
表1 12 个与HCC 预后有关自噬相关lncRNA
Table 1 The 12 autophagy-related lncRNAs significantly associated with the prognosis of HCC

lncRNA KM B SE HR 95% CI P CYTOR 0.002 0.064 0.018 1.067 1.029~1.105<0.001 DANCR 0.014 0.028 0.009 1.029 1.011~1.047 0.001 LINC00864 0.046 -0.372 0.182 0.689 0.482~0.986 0.041 LINC01138 0.002 0.472 0.127 1.604 1.250~2.057<0.001 LINC01871 0.004 -0.146 0.069 0.864 0.754~0.989 0.035 LINC02362 0.014 -0.106 0.043 0.899 0.825~0.980 0.016 LUCAT1 0.023 0.282 0.067 1.325 1.163~1.510<0.001 MAPKAPK5-AS1 0.002 0.104 0.041 1.11 1.025~1.201 0.01 NRAV<0.001 0.197 0.053 1.218 1.099~1.350<0.001 NRSN2-AS1 0.029 0.24 0.096 1.272 1.054~1.535 0.012 PSMB8-AS1 0.009 -0.089 0.042 0.915 0.843~0.993 0.033 TMEM220-AS1 0.002 -0.278 0.095 0.757 0.629~0.911 0.003

图1 HCC 中12 个lncRNA 与自噬基因共表达的网络图(lncRNA 为红色节点,自噬基因为蓝色节点)
Figure 1 Network of the 12 lncRNAs with the co-expressed autophagy genes in HCC (the red nodes indicating the lncRNAs,the blue nodes indicating the autophagy genes)

图2 训练集中12 个lncRNA 的生存曲线图
Figure 2 Survival curves for the 12 prognostic lncRNAs in the train dataset

图3 12 个lncRNAs 与自噬基因关系及对HCC 患者预后的影响
Figure 3 The relationship between 12 lncRNAs and autophagy genes and its effect on the prognosis of the HCC patients
表2 训练集中12 个lncRNA 多因素Cox 回归分析
Table 2 Multivariate Cox regression analysis of the 12 lncRNAs in the train dataset
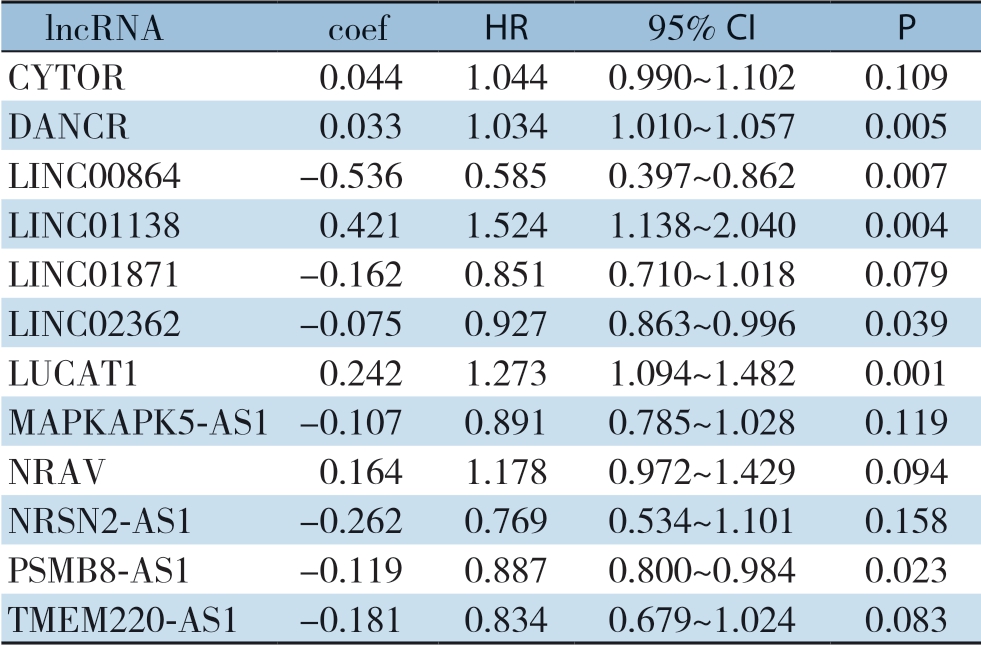
lncRNA coef HR 95% CI P CYTOR 0.044 1.044 0.990~1.102 0.109 DANCR 0.033 1.034 1.010~1.057 0.005 LINC00864 -0.536 0.585 0.397~0.862 0.007 LINC01138 0.421 1.524 1.138~2.040 0.004 LINC01871 -0.162 0.851 0.710~1.018 0.079 LINC02362 -0.075 0.927 0.863~0.996 0.039 LUCAT1 0.242 1.273 1.094~1.482 0.001 MAPKAPK5-AS1 -0.107 0.891 0.785~1.028 0.119 NRAV 0.164 1.178 0.972~1.429 0.094 NRSN2-AS1 -0.262 0.769 0.534~1.101 0.158 PSMB8-AS1 -0.119 0.887 0.800~0.984 0.023 TMEM220-AS1 -0.181 0.834 0.679~1.024 0.083

图4 训练集中HCC 患者的自噬相关lncRNA 风险评分分析
Figure 4 Risk score analysis of the autophagy-related lncRNAs in train dataset
A:自噬相关lncRNA 信号的低分组与高分组;B:HCC 患者存活状态及生存时间;C:HCC 患者中12 个lncRNA 表达的热图
A:The low score group and high score group of the autophagy-related lncRNA signals;B:The survival status and duration of the HCC patients;C:Heatmap of the expressions of the 12 key lncRNAs in HCC patients
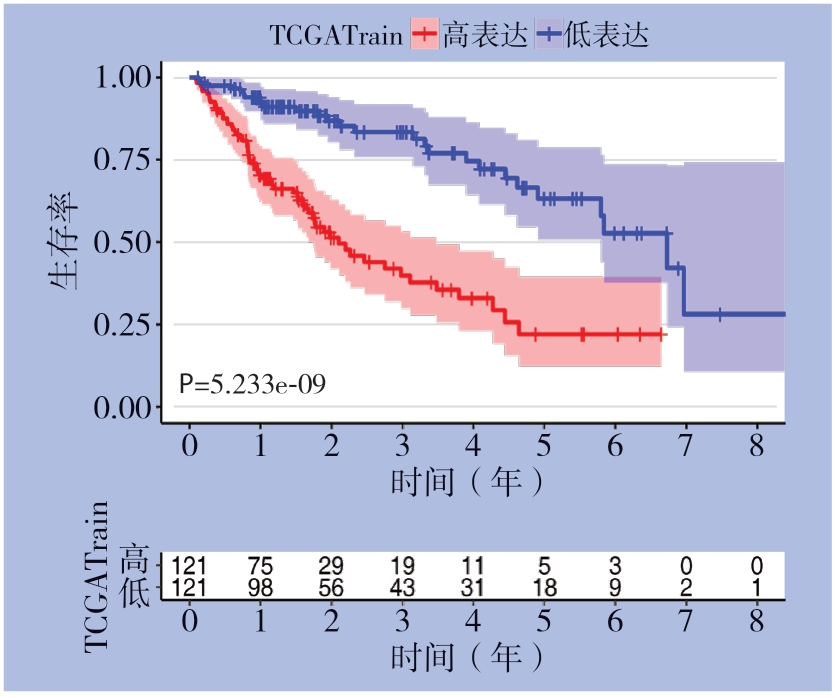
图5 训练集中不同风险评分HCC 患者的生存曲线
Figure 5 Survival curves of the HCC patients with different risk scores in the train dataset
表3 训练集HCC 患者临床特征及风险评分的多因素Cox回归分析
Table 3 Multivariate Cox regression analysis of characteristics and risk score of the HCC patients in the train dataset
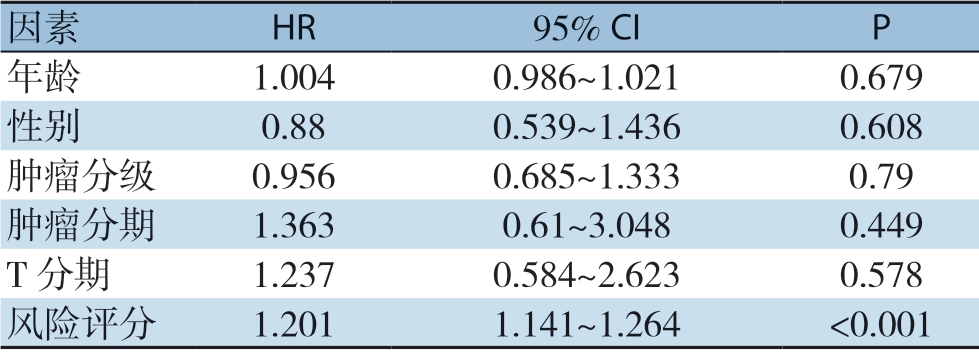
因素 HR 95% CI P年龄 1.004 0.986~1.021 0.679性别 0.88 0.539~1.436 0.608肿瘤分级 0.956 0.685~1.333 0.79肿瘤分期 1.363 0.61~3.048 0.449 T 分期 1.237 0.584~2.623 0.578风险评分 1.201 1.141~1.264<0.001
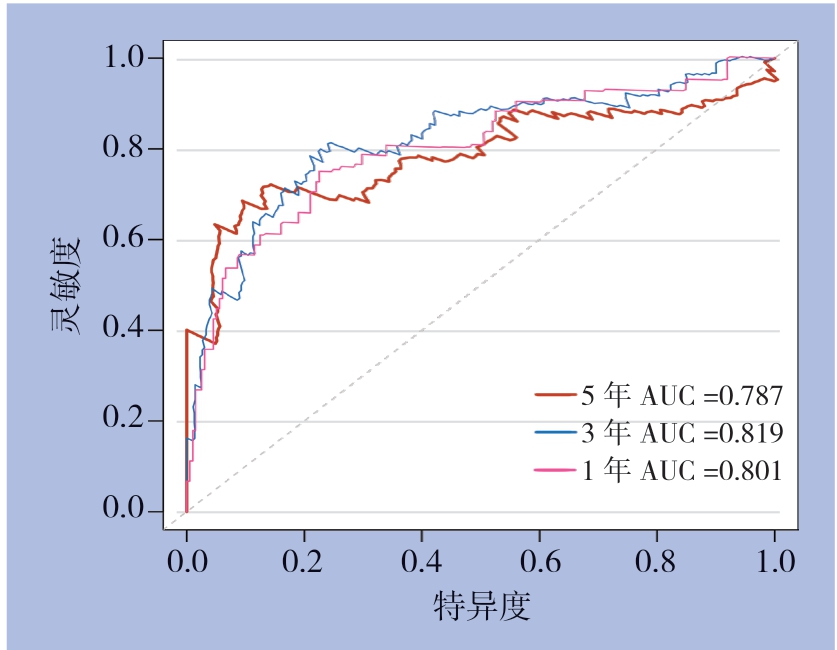
图6 训练集中风险模型的1、3、5年生存的时间依赖性ROC 曲线
Figure 6 Time-dependent ROC curves of the risk model for the 1-,3- and 5-year survival in train dataset
2.4 预后模型与HCC 患者的临床病理特征的关系
随后分析了12-自噬相关lncRNA风险评分在年龄、性别、分级、分期等方面的临床价值。结果显示,肿瘤分级、肿瘤分期和T分期越高,患者风险评分越高,提示该12-自噬相关lncRNA模型可能与HCC的进展有关;而在不同年龄和性别间无统计学差异(均P>0.05)(表4)。
表4 预后模型风险评分与患者临床因素的关系
Table 4 The relationship between risk score and clinical factors in patients train dataset
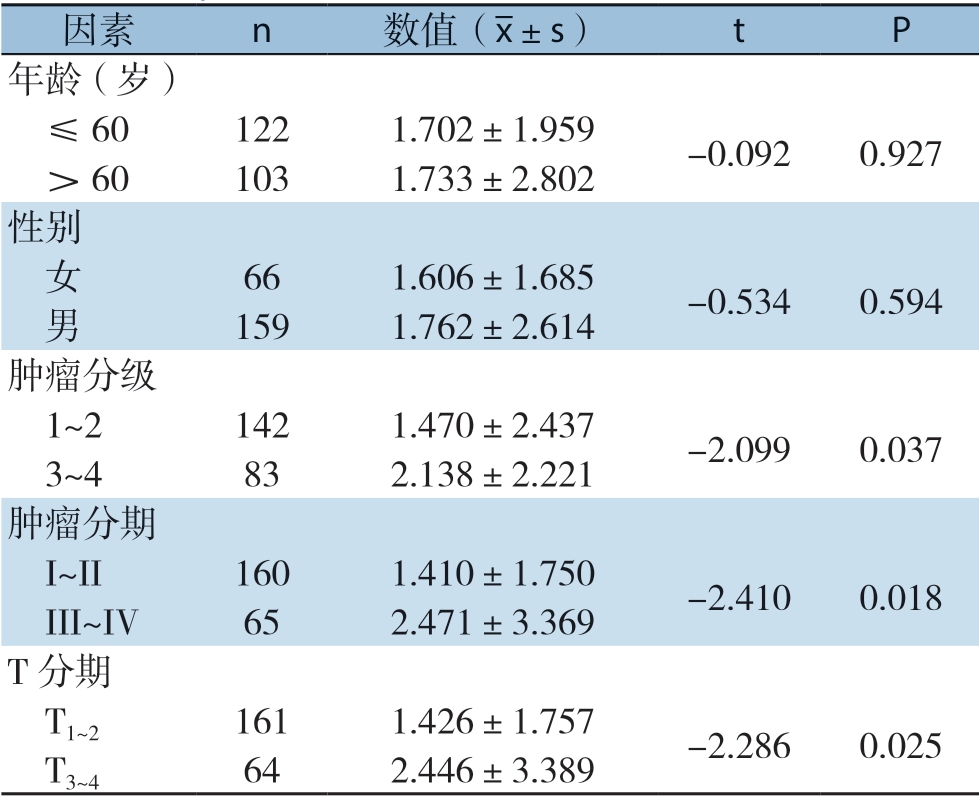
因素 n 数值(images/BZ_40_1331_2693_1363_2730.png±s) t P年龄(岁) ≤60 122 1.702±1.959 -0.092 0.927 >60 103 1.733±2.802性别 女 66 1.606±1.685 -0.534 0.594 男 159 1.762±2.614肿瘤分级 1~2 142 1.470±2.437 -2.099 0.037 3~4 83 2.138±2.221肿瘤分期 I~II 160 1.410±1.750 -2.410 0.018 III~IV 65 2.471±3.369 T 分期 T1~2 161 1.426±1.757 -2.286 0.025 T3~4 64 2.446±3.389
2.5 内部验证集中的验证
使用相同coef值在内部验证集(n=100)中进一步验证这些结果。根据风险评分的中位值将这些患者分为高风险和低风险组。与从训练集得出的结果一致,高风险患者的中位OS比低风险患者的中位OS短(P=0.0236)(图7)。所构建的lncRNA模型在内部验证集中预测患者1、3、5年生存的时间依赖性ROC曲线的AUC分别为0.694、0.733和0.746(图8)。
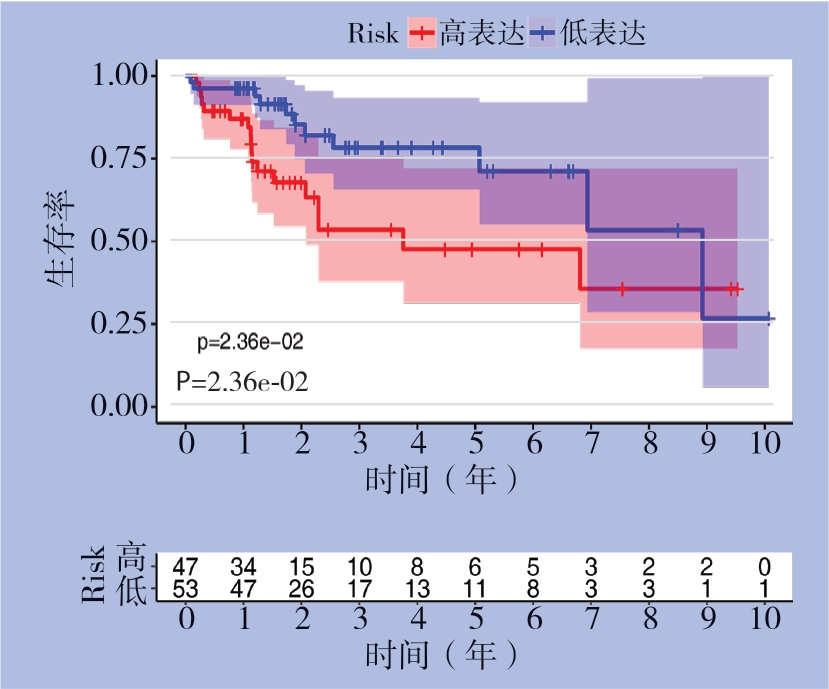
图7 验证集中不同风险评分HCC 患者的生存曲线
Figure 7 Survival curves of the HCC patients with different risk scores in the validation dataset
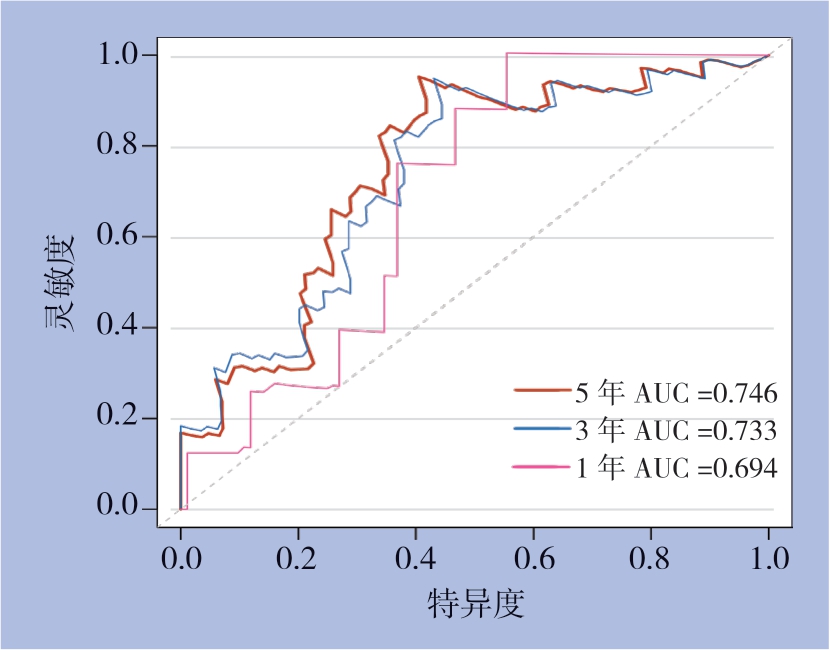
图8 验证集中预后模型的1、3、5年生存的时间依赖性ROC 曲线
Figure 8 Time-dependent ROC curves of the risk model for the 1-,3- and 5-year survival in the validation dataset
3 讨 论
随着高通量测序技术的成熟,通过检测lncRNA表达的改变来预测HCC的预后的研究也越来,例如UCA1[24]和HOTAIR[25]。已有研究[26]表明可以使用多个自噬相关lncRNA构建模型有效的预测神经胶质瘤患者的预后。目前为止,还没有可以预测HCC患者生存的自噬相关lncRNA模型。因此,有必要建立相关预后模型来预测HCC患者的预后。
在本研究中,通过TCGA数据库中HCC数据集探讨HCC患者自噬相关lncRNA的预后作用。本研究通过lncRNA-自噬基因共表达网络鉴定了347个自噬相关lncRNA。随后筛选出12个最具预后意义的自噬相关lncRNA。在12种自噬相关的lncRNA中,CYTOR、DANCR、LINC01138、LUCAT1、MAPKAPK5-AS1、NRAV和NRSN2-AS1的高表达与患者总生存期成负相关,而LINC01871、LINC00864、LINC02362、TMEM220-AS1 和PSMB8-AS1 的高表达与患者的总生存期成正相关。在先前的研究中已经报道了这些基因中的大多数在HCC 或其他恶性肿瘤的预后密切相关。LINC01138[27-28]在HCC 组织中表达显着增加,并且与HCC 的临床病理特征呈正相关,可能是HCC患者预后预测的潜在有价值的候选标志物。DANCR[29]被证明在HCC组织中明显过表达,并与HCC患者的预后呈负相关,且在体外和体内促进HCC细胞中索拉非尼的耐药性。MAPKAPK5-AS1[30]在HCC中与总体存活率显着相关,有可能成为HCC的预后因子。LUCAT1[31]在HCC组织中高表达,且其与患者年龄、肿瘤组织学分级、肿瘤T分期和生存状态显著相关,是HCC患者生存不良的独立危险因素。本研究和这些有类似的结果。
使用12-自噬相关lncRNA构建预后模型,计算风险评分,分析风险评分与临床病理特征及预后的关系。结果显示,在训练集中风险评分与HCC患者肿瘤分级、肿瘤分期和T分期有关(P<0.05),在较高的肿瘤分级、分期中,患者的风险评分趋于增加,表明该lncRNA模型可能与HCC的进展有关。通过多变量Cox回归分析,该风险预后模型是与影响HCC患者预后的独立危险因素(P<0.001)。该风险模型对训练集HCC患者的1、3、5年生存的时间依赖性ROC曲线的AUC分别为0.801,0.819和0.787,同时在验证集中同样具有较好的预测能力,其1、3、5年生存的时间依赖性ROC曲线的AUC分别为0.694、0.733和0.746。本研究构建的自噬lncRNA预后模型在内部验证集中同样具有较好的灵敏度和特异性。根据这些结果,该12-自噬相关lncRNA模型为HCC患者的预后提供可靠的预测。而且12-自噬相关lncRNA是潜在的自噬相关治疗靶点,对于个性化治疗方案的制定也可能具有参考价值。
本研究仍存在局限性,首先,这是一项回顾性研究,研究数据源自于TCGA数据库,因此可能产生一些误差。其次,该预后模型仍有必要在其他独立的队列中进一步进行验证,以明确该模型的稳定性,对于该模型基因中未被证实功能性实验证实与HCC预后相关的基因需要进一步实验揭示其潜在作用机制。
通过构建自噬基因-lncRNA共表达网络,鉴定了12个自噬相关的lncRNA的预测模型,该模型对预测HCC患者1、3、5年生存率均有较好的灵敏度及特异性。虽然本研究中12-自噬相关lncRNA风险预后模型可以较准确的预测HCC患者的预后,但仍需要通过前瞻性实验来进一步验证。
[1]Bray F,Ferlay J,Soerjomataram I,et al.Global cancer statistics 2018:GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries[J].CA Cancer J Clin,2018,68(6):394-424.doi:10.3322/caac.21492.
[2]陈世发,赵礼金.肝癌发生发展机制的研究进展及其治疗现状[J].中国普通外科杂志,2018,27(7):910-923.doi:10.3978/j.issn.1005-6947.2018.07.016.
Chen SF,Zhao LJ.Research progress on mechanisms for occurrence of liver cancer and its treatment status[J].Chinese Journal of General Surgery,2018,27(7):910-923.doi:10.3978/j.issn.1005-6947.2018.07.016.
[3]Song X,Du R,Gui H,et al.Identification of potential hub genes related to the progression and prognosis of hepatocellular carcinoma through integrated bioinformatics analysis[J].Oncol Rep,2020,43(1):133-146.doi:10.3892/or.2019.7400.
[4]乐琪,朱同恩,莫志远,等.原发性肝癌患者手术切除术后早期复发影响因素分析[J].中国普通外科杂志,2019,28(1):18-23.doi:10.7659/j.issn.1005-6947.2019.01.003.
Le Q,Zhu TE,Mo ZY,et al.Analysis of factors for early recurrence of patients with hepatocellular carcinoma after resection[J].Chinese Journal of General Surgery,2019,28(1):18-23.doi:10.7659/j.issn.1005-6947.2019.01.003.
[5]Maluccio M,Covey A.Recent progress in understanding,diagnosing,and treating hepatocellular carcinoma[J].CA Cancer J Clin,2012,62(6):394-399.doi:10.3322/caac.21161.
[6]Levy J,Towers CG,Thorburn A.Targeting autophagy in cancer[J].Nat Rev Cancer,2017,17(9):528-542.doi:10.1038/nrc.2017.53.
[7]Luo L,Qin ZH.Autophagy,Aging,and Longevity[J].Adv Exp Med Biol,2019,1206:509-525.doi:10.1007/978-981-15-0602-4_24.
[8]Kocaturk NM,Akkoc Y,Kig C,et al.Autophagy as a molecular target for cancer treatment[J].Eur J Pharm Sci,2019,134:116-137.doi:10.1016/j.ejps.2019.04.011.
[9]Rajendran P,Alzahrani A M,Hanieh HN,et al.Autophagy and senescence:A new insight in selected human diseases[J].J Cell Physiol,2019,234(12):21485-21492.doi:10.1002/jcp.28895.
[10]Gual P,Gilgenkrantz H,Lotersztajn S.Autophagy in chronic liver diseases:the two faces of Janus[J].Am J Physiol Cell Physiol,2017,312(3):C263-273.doi:10.1152/ajpcell.00295.2016.
[11]Deng S,Shanmugam MK,Kumar AP,et al.Targeting autophagy using natural compounds for cancer prevention and therapy[J].Cancer,2019,125(8):1228-1246.doi:10.1002/cncr.31978.
[12]Cirone M,Gilardini Montani MS,et al.Autophagy manipulation as a strategy for efficient anticancer therapies:possible consequences[J].J Exp Clin Cancer Res,2019,38(1):262.doi:10.1186/s13046-019-1275-z.
[13]White E,DiPaola R S.The double-edged sword of autophagy modulation in cancer[J].Clin Cancer Res,2009,15(17):5308-5316.doi:10.1158/1078-0432.CCR-07-5023.
[14]胡若冰,杨玉秀,李修岭,等.氯喹对肝癌细胞体内外生长的影响及其与自噬的关系[J].中国普通外科杂志,2020,29(1):61-68.doi:10.7659/j.issn.1005-6947.2020.01.008.
Hu RB,Yang YX,Li XL,et al.Effect of chloroquine on growth of liver cancer cells and its relationship with autophagy[J].Chinese Journal of General Surgery,2020,29(1):61-68.doi:10.7659/j.issn.1005-6947.2020.01.008.
[15]Lee YG,Jeon TI.Modulation of the Autophagy-lysosomal Pathway in Hepatocellular Carcinoma Using Small Molecules[J].Molecules,2020,25(7):1580.doi:10.3390/molecules25071580.
[16]Kiruthiga C,Devi KP,Nabavi SM,et al.Autophagy:A Potential Therapeutic Target of Polyphenols in Hepatocellular Carcinoma[J].Cancers (Basel),2020,12(3):562.doi:10.3390/cancers12030562.
[17]Li P,Du Q,Cao Z,et al.Interferon-γ induces autophagy with growth inhibition and cell death in human hepatocellular carcinoma (HCC) cells through interferon-regulatory factor-1 (IRF-1)[J].Cancer Lett,2012,314(2):213-222.doi:10.1016/j.canlet.2011.09.031.
[18]Lin A,Li C,Xing Z,et al.The LINK-A lncRNA activates normoxic HIF1α signalling in triple-negative breast cancer[J].Nat Cell Biol,2016,18(2):213-224.doi:10.1038/ncb3295.
[19]Rachagani S,Macha MA,Heimann N,et al.Clinical implications of miRNAs in the pathogenesis,diagnosis and therapy of pancreatic cancer[J].Adv Drug Deliv Rev,2015,81:16-33.doi:10.1016/j.addr.2014.10.020.
[20]Guo Q,Cheng Y,Liang T,et al.Comprehensive analysis of lncRNA-mRNA co-expression patterns identifies immuneassociated lncRNA biomarkers in ovarian cancer malignant progression[J].Sci Rep,2015,5:17683.doi:10.1038/srep17683.
[21]Pang W,Lian F Z,Leng X,et al.Microarray expression profiling and co-expression network analysis of circulating lncRNAs and mRNAs associated with neurotoxicity induced by BPA[J].Environ Sci Pollut Res Int,2018,25(15):15006-15018.doi:10.1007/s11356-018-1678-y.
[22]Yang L,Zhang X,Li H,et al.The long noncoding RNA HOTAIR activates autophagy by upregulating ATG3 and ATG7 in hepatocellular carcinoma[J].Mol Biosyst,2016,12(8):2605-2612.doi:10.1039/c6mb00114a.
[23]Jing Z,Ye X,Ma X,et al.SNGH16 regulates cell autophagy to promote Sorafenib Resistance through suppressing miR-23b-3p via sponging EGR1 in hepatocellular carcinoma[J].Cancer Med,2020,9(12):4324-4338.doi:10.1002/cam4.3020.
[24]Zhang Z,Li JZ,Wei ZW,et al.Correlation between expression levels of lncRNA UCA1 and miR-18a with prognosis of hepatocellular cancer[J].Eur Rev Med Pharmacol Sci,2020,24(7):3586-3591.doi:10.26355/eurrev_202004_20820.
[25]Gong X,Zhu Z.Long Noncoding RNA HOTAIR Contributes to Progression in Hepatocellular Carcinoma by Sponging miR-217-5p[J].Cancer Biother Radiopharm,2020,35(5):387-396.doi:10.1089/cbr.2019.3070.
[26]Luan F,Chen W,Chen M,et al.An autophagy-related long noncoding RNA signature for glioma[J].FEBS Open Bio,2019,9(4):653-667.doi:10.1002/2211-5463.12601.
[27]Li Z,Zhang J,Liu X,et al.The LINC01138 drives malignancies via activating arginine methyltransferase 5 in hepatocellular carcinoma[J].Nat Commun,2018,9(1):1572.doi:10.1038/s41467-018-04006-0.
[28]Jiang H,Shi X,Ye G,et al.Up-regulated Long Non-Coding RNA DUXAP8 Promotes Cell Growth Through Repressing Krüppel-like Factor 2 Expression in Human Hepatocellular Carcinoma[J].Onco Targets Ther,2019,12:7429-7436.doi:10.2147/OTT.S214336.
[29]Liu Y,Chen L,Yuan H,et al.LncRNA DANCR Promotes Sorafenib Resistance via Activation of IL-6/STAT3 Signaling in Hepatocellular Carcinoma Cells[J].Onco Targets Ther,2020,13:1145-1157.doi:10.2147/OTT.S229957.
[30]Zhang J,Fan D,Jian Z,et al.Cancer Specific Long Noncoding RNAs Show Differential Expression Patterns and Competing Endogenous RNA Potential in Hepatocellular Carcinoma[J].PLoS One,2015,10(10):e0141042.doi:10.1371/journal.pone.0141042.
[31]Jiao Y,Li Y,Ji B,et al.Clinical Value of lncRNA LUCAT1 Expression in Liver Cancer and its Potential Pathways[J].J Gastrointestin Liver Dis,2019,28(4):439-447.doi:10.15403/jgld-356.
